El virus de Lassa: estudios y posibles dianas terapéuticas
La fiebre de Lassa es una enfermedad vírica hemorrágica aguda que no tiene tratamiento ni vacunas aprobadas para humanos y que se transmite por contacto con alimentos o utensilios domésticos contaminados con orina o heces de roedores. El catedrático de Bioquímica y Biología Molecular de la Universidad Miguel Hernández (UMH) José Villalaín ha realizado un análisis virtual del virus que produce esta enfermedad y ha comprobado que ciertas partes de su estructura serían buenas candidatas para el diseño de fármacos que eviten la infección. En África Occidental se producen cada año entre 100.000 y 300.000 contagios de virus de Lassa.
Los virus no pueden multiplicarse por sí solos y necesitan infectar células de otro organismo para hacerlo. El virus de Lassa entra en las células huésped cuando las proteínas de su envuelta se unen a los receptores moleculares del endosoma y el virus completo es transportado al interior de la célula. En concreto, este anclaje se produce mediante los complejos de glicoproteínas de la superficie del virus. Estudiar este complejo y sus componentes, explica el profesor Villalaín, es un paso previo importante para la creación de fármacos que eviten la infección y paren el ciclo vital del virus.
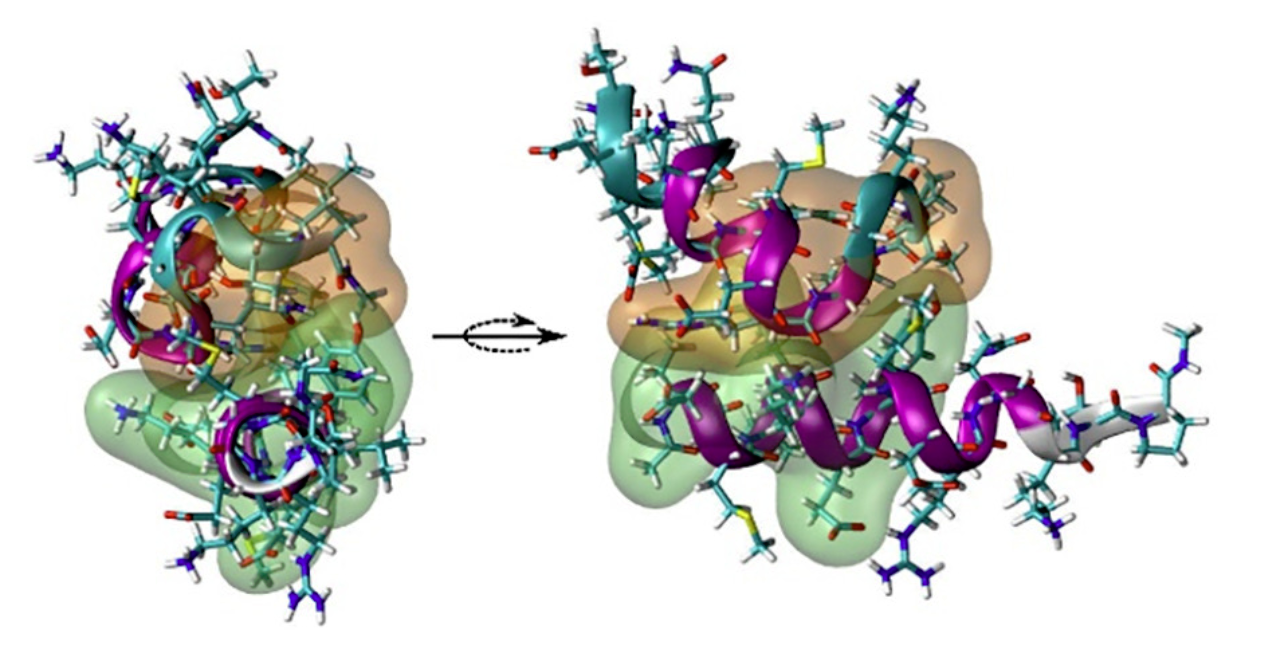
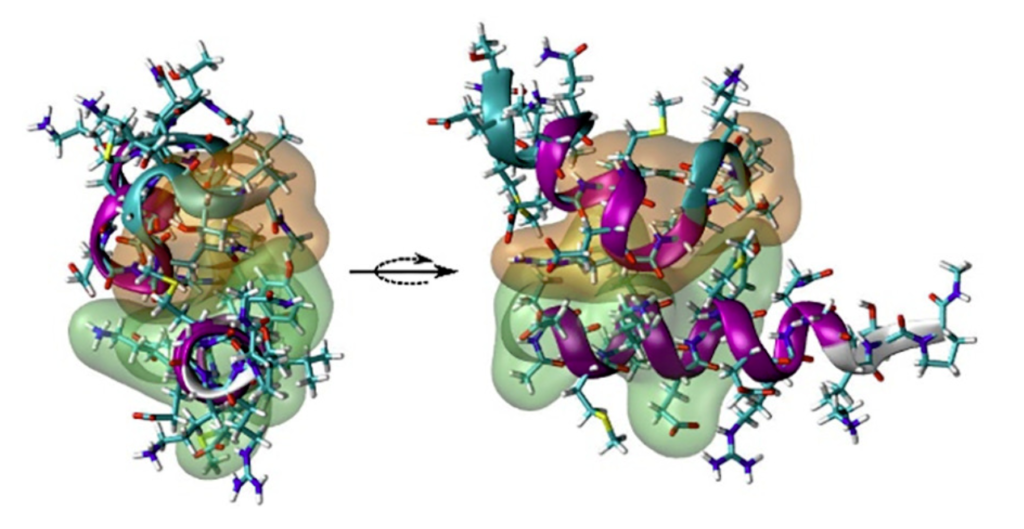
En el caso particular del virus de Lassa, estas proteínas de anclaje tienen dos componentes, llamados NFP (que se da en el virus del sida y la gripe) e IFL (característico del virus del Ébola). Solo los coronavirus tienen, como el agente causante del COVID, tienen, también, este tipo de proteínas de anclaje. En estos componentes y en los dominios transmembrana (TM) y la región externa de la membrana proximal (MPER) se ha centrado el investigador del Instituto de Investigación, Desarrollo e Innovación en Biotecnología Sanitaria de Elche (IDiBE) de la UMH.
Estudios y simulaciones para analizar el virus
Mediante una simulación por ordenador, el profesor Villalaín ha demostrado la capacidad de los dominios NFP/IFL, MPER y TM del virus de interaccionar con la membrana de la célula huésped y, por tanto, la posibilidad de obtener inhibidores de la entrada del virus, es decir, impedir la infección viral causante de la fiebre hemorrágica Lassa.
Este estudio se ha realizado mediante dinámica molecular, un proceso de ‘simulación virtual’ de la interacción entre proteínas. Esto es particularmente útil en el caso del virus de Lassa, ya que se considera un agente biológico muy peligroso y su estudio con células reales requiere un nivel de bioseguridad 4, como el Ébola, la viruela o el hantavirus.
Sin embargo, analizar virtualmente la estructura de un virus requiere tanta potencia informática que ha de utilizarse un conjunto de ordenadores para llevar a cabo el experimento. En estas simulaciones, se puede ver cómo se comporta cada átomo que compone las proteínas del virus, con lo que se pueden estudiar en detalle tanto el proceso de infección como un posible tratamiento. Para este tipo de investigaciones, la UMH cuenta con un clúster de computación científica, un grupo de ordenadores unidos mediante una red de alta velocidad, promovido por el Vicerrectorado de Tecnologías de la Información y gestionado por el Servicio de Innovación y Planificación Tecnológica.
El objetivo de estudiar esta enfermedad
El profesor Villalaín explica que la fiebre hemorrágica de Lassa es endémica en algunos países de África y la Organización Mundial de la Salud considera que es una de las cinco enfermedades infecciosas más importantes que hay que vigilar cara al futuro por su peligrosidad. Como la mayoría de los casos son leves o asintomáticos (80%) la letalidad global es de aproximadamente el 1%, pero en los casos graves, que precisan hospitalización, la letalidad llega al 15%, y en algunas epidemias llegará hasta el 50%.
El trabajo llevado a cabo en la UMH es fundamental para poder definir dianas terapéuticas y de esta manera poder obtener moléculas antivirales contra este virus y otros similares. Los resultados, publicados en la revista Biochimica Biophysica Acta Biomembranes, demuestran la capacidad de interacción con las biomembranas de los domínios NFP/IFL, MPER y TM de la proteína GP2 del virus Lassa.
Enlace a la publicación: https://doi.org/10.1016/j.bbamem.2022.184031
Si deseas leer más artículos como este, puedes entrar en UMH Sapiens.