Descifraron el código de las sorprendentes estructuras de las proteínas
Baker, Hassabis y Jumper, Nobel de Química por descifrar la estructura de las proteínas con herramientas de inteligencia artificial
⬛ SINC / Ángeles Gallar
“Hoy informamos sobre el desarrollo y las aplicaciones iniciales de RoseTTAFold, una herramienta de software que utiliza el aprendizaje profundo para predecir de forma rápida y precisa las estructuras de las proteínas basándose en información limitada. Sin la ayuda de este tipo de software, puede llevar años de trabajo de laboratorio determinar la estructura de una sola proteína. Con RoseTTAFold, se puede calcular la estructura de una proteína en tan solo diez minutos en una sola computadora para juegos. Este trabajo fue dirigido por el investigador postdoctoral del laboratorio Baker, Minkyung Baek, Ph.D.”
Así se anunciaba el primer software capaz de predecir las estructuras tridimensionales de las proteínas el 15 de julio de 2021. Apenas tres años después, el científico que lideró el desarrollo de RoseTTAFold, David Baker (EE.UU., 1962), ha recibido la llamada de la Academia Sueca de las Ciencias. Este programa revolucionario comparte el Premio Nobel de Química del 2024 al 50% con el británico Demis Hassabis (1976) y el también estadounidense John M. Jumper (1985), ambos de la empresa DeepMind de Google, que lograron predecir las estructuras proteicas utilizando Inteligencia Artificial en 2022. Baker, Hassabis y Jumper fueron también premiados por la Fundación BBVA Fronteras del Conocimiento en la categoría de Biomedicina en 2023.

La vida no podría existir sin proteínas. “Que ahora podamos predecir las estructuras de las proteínas y diseñar nuestras propias proteínas confiere el mayor beneficio a la humanidad”, dice el comunicado del Comité del Premio Nobel de Química.
Sin ir más lejos, en la Universidad Miguel Hernández de Elche (UMH) se utilizan las simulaciones virtuales de proteínas para predecir los efectos terapéuticos de vacunas y de otros medicamentos frente a infecciones virales, o para predecir los efectos de los disruptores endocrinos en las moléculas de un organismo vivo, entre muchas otras investigaciones.
Proteínas sintéticas con múltiples aplicaciones
“Uno de los descubrimientos reconocidos este año se refiere a la construcción de proteínas espectaculares. El otro consiste en hacer realidad un sueño de 50 años: predecir las estructuras de las proteínas a partir de sus secuencias de aminoácidos. Ambos descubrimientos abren enormes posibilidades”, ha declarado Heiner Linke, presidente del Comité Nobel de Química.
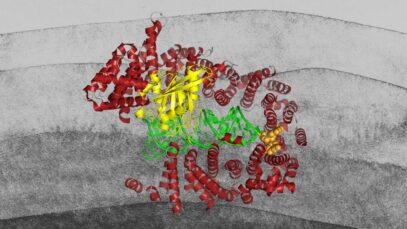
Las proteínas suelen estar formadas por 20 aminoácidos diferentes, que pueden describirse como los componentes básicos de la vida. En 2003, Baker consiguió utilizar estos bloques para diseñar una nueva proteína que no se parecía a ninguna otra. Desde entonces, su grupo de investigación ha producido una creación proteínica imaginativa tras otra, incluidas proteínas que pueden utilizarse como fármacos, vacunas, nanomateriales y sensores diminutos, mediante RoseTTAFold y nuevas versiones del programa.
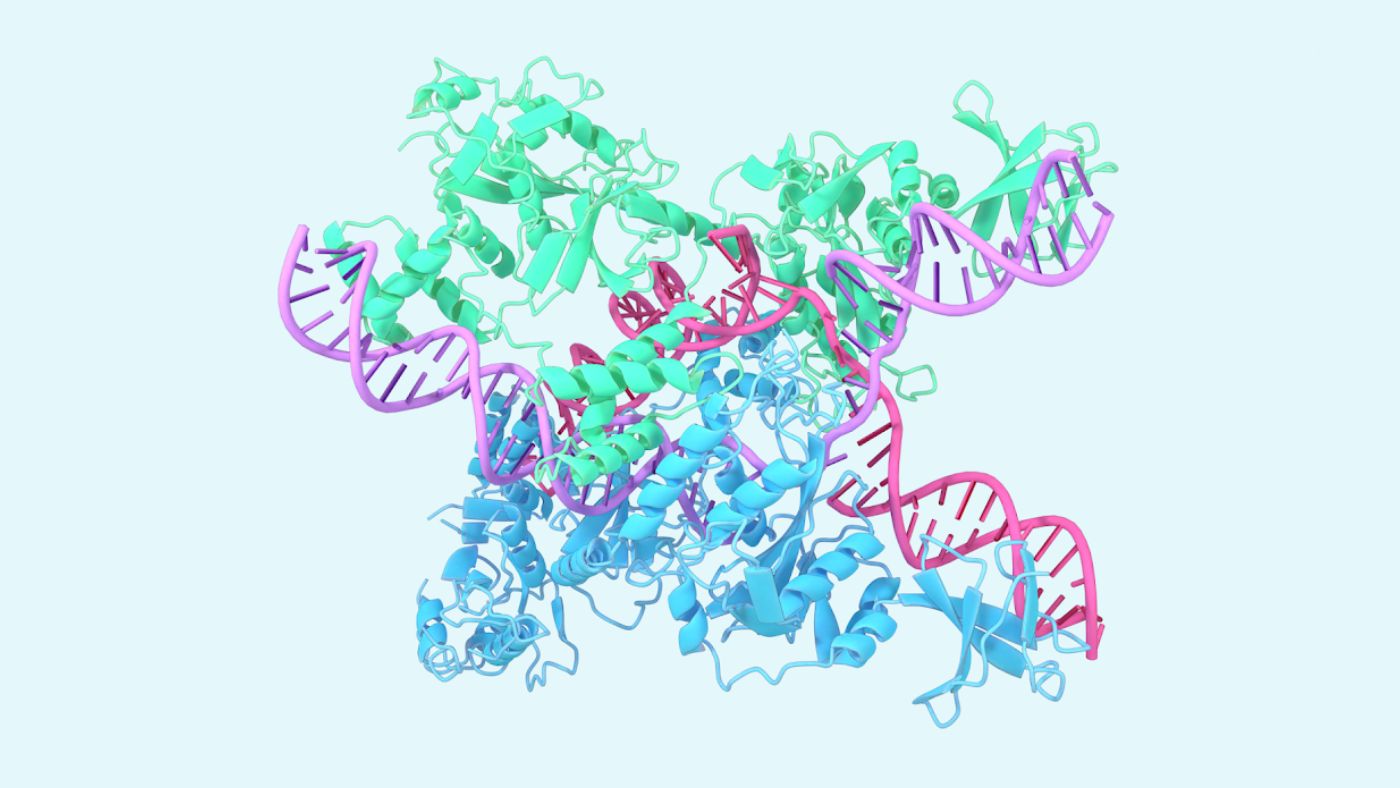
El segundo descubrimiento se refiere a la predicción de estructuras proteicas. En las proteínas, los aminoácidos están unidos en largas cadenas que se pliegan para formar una estructura tridimensional, decisiva para la función de la proteína.
Desde los años 70, los investigadores habían intentado predecir las estructuras de las proteínas a partir de secuencias de aminoácidos, pero resultaba notoriamente difícil. Sin embargo, hace cuatro años se produjo un avance asombroso.
En 2020, Hassabis y Jumper presentaron AlphaFold2, con este modelo de IA han podido predecir la estructura de prácticamente los los 200 millones de proteínas identificadas por la ciencia.
En 2020, Hassabis y Jumper presentaron un modelo de IA llamado AlphaFold2. Con su ayuda, han podido predecir la estructura de prácticamente todos los 200 millones de proteínas que los investigadores han identificado. Desde su descubrimiento, AlphaFold2 ha sido utilizado por más de dos millones de personas de 190 países. Entre un sinfín de aplicaciones científicas, los investigadores pueden ahora comprender mejor la resistencia a los antibióticos y crear imágenes de enzimas capaces de descomponer el plástico.
Artículos relacionados:
- Baek, M., DiMaio, F., Anishchenko, I., Dauparas, J., Ovchinnikov, S., Lee, G. R., … & Baker, D. (2021). Accurate prediction of protein structures and interactions using a three-track neural network. Science, 373(6557), 871-876. https://www.science.org/doi/10.1126/science.abj8754
- Jumper, J., Evans, R., Pritzel, A. et al. Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 (2021). https://doi.org/10.1038/s41586-021-03819-2